使用机器学习算法深入了解细胞子集组成
客观的
介绍一个20标记面板,适合在CytoFLEX LX流式细胞仪上采集。将探索在Cytobank平台上使用viSNE、FlowSOM和SPADE对数据进行可视化。将展示使用Kaluza分析的手动选通策略,并将其与Cytobank平台上FlowSOM无监督聚类识别的子集进行比较。1,2其中许多算法的无监督性质减少了手动选通已知亚群可能引入的偏差,并使研究人员能够识别意外表型。与手动选通相比,使用机器学习算法实现高维数据集的详尽分析所需的动手时间减少了这是一个额外的优势。
为了生成本应用笔记中使用的数据,在使用Versalyse进行红细胞裂解之前,血液样本用20色抗体鸡尾酒进行染色(见表1),按照标准程序(部件编号)IM3648).在6激光上采集染色样本CytoFLEX LX流式细胞仪. 以下描述的过滤器配置已用于确保每种染料的最佳检测。
表1。使用CytoFLEX LX流式细胞仪进行20标记物免疫分型的面板组成。
使用逻辑扩展进行补偿和数据转换卡鲁扎分析软件并将补偿后的逻辑转换数据导出到细胞库平台使用Kaluza细胞库插件。Kaluza分析也用于双轴门控和手动群体识别。
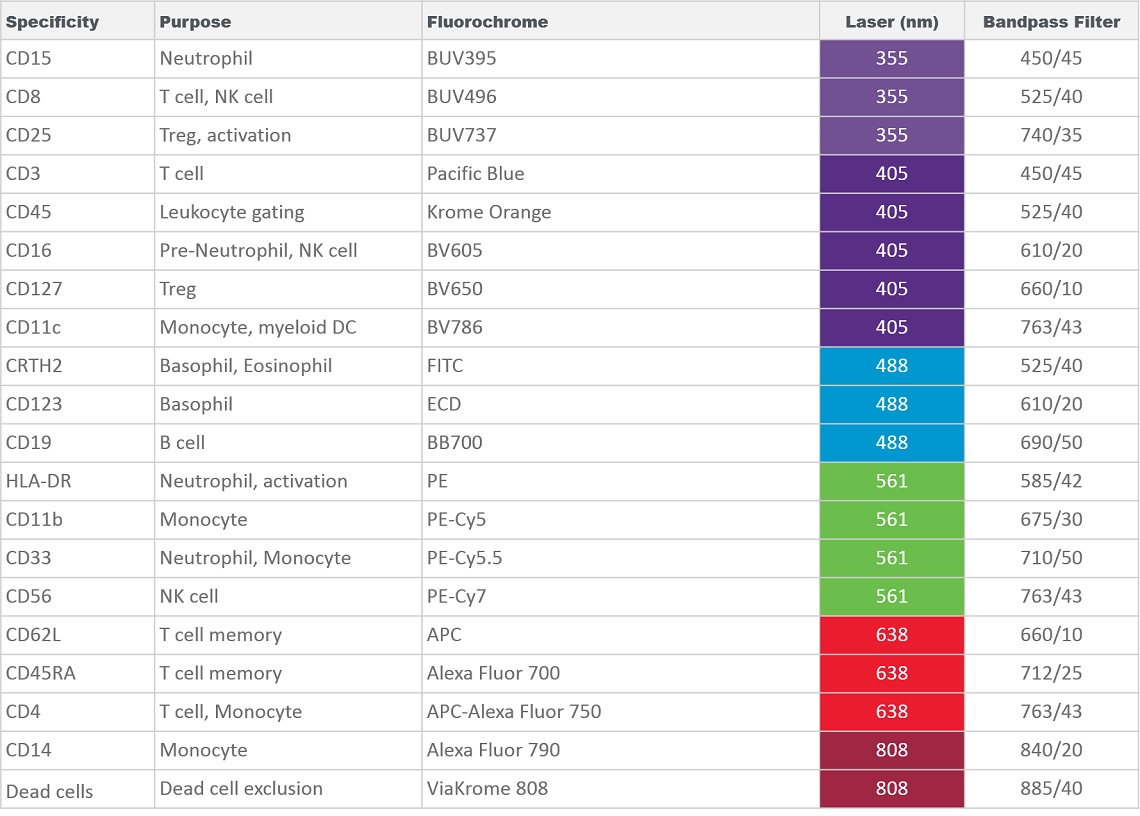
在为机器学习辅助的数据分析做准备时,碎片、双峰,通常还有死细胞或其他不想要的事件都会被移除。这些事件不会为下游分析增加信息,如果不适当地识别和排除,可能会对数据显示产生负面影响,并混淆统计结果。根据所需的数据可视化和研究问题,在感兴趣的总体上预先进行进一步分析可能很有价值(图1)。
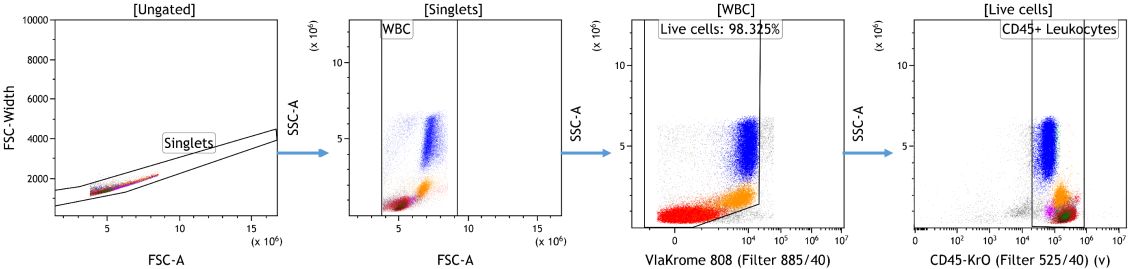
图1。Kaluza分析中的数据清理.根据前向散射信号面积与高度排除双峰,然后根据前向和侧散射特性对白细胞进行门控。细胞阴性的ViaKrome 808(零件号C36628)根据CD45的表达,被鉴定为活细胞,并进一步分类为白细胞。使用Kaluza分析软件分析数据。绘图仅用于说明目的。
为了手动识别人类外周血样本中的细胞亚群,我们使用Kaluza Analysis软件建立了一种基于先前公布的标记表达模式知识的门控策略。3.
 图2。主要白细胞亚群的知识驱动识别.数据分析使用Kaluza分析软件。绘图仅供说明之用。
图2。主要白细胞亚群的知识驱动识别.数据分析使用Kaluza分析软件。绘图仅供说明之用。
使用机器学习工具,可以在没有偏见和独立于先验知识的情况下识别细胞子集。viSNE等降维算法能够将高维数据集中包含的信息可视化到单个2D图1中。FlowSOM等聚类算法能够根据标记表达的相似性自动识别和分组相似的细胞。2.
为了进一步分析数据集,viSNE用于所有选通标记的降维,这些标记也用于图2所示的手动选通步骤。这允许在单个2D图中显示这11个标记(CD45、CRTH2、CD123、CD15、CD14、CD16、CD56、CD3、CD4、CD8、CD19)中包含的信息。viSNE是一种将高维数据简化为二维的方法,从而实现快速探索性数据分析和复杂结果的可视化。对于细胞仪数据,这可能有助于将事件/细胞分类为生物群体。表型相似的细胞彼此靠近,形成一个岛。所得viSNE图的等高线图如图3A所示。
降维后,进行FlowSOM分析,将细胞自动聚类为12个所谓的元聚类。在群体定义标记上运行FlowSOM,并在viSNE地图上显示结果聚类数据,可促进质量评估。如果需要进一步迭代调整算法运行设置以优化结果,此可视化可能有助于比较不同的运行,并为聚类数据的分析提供起点。
在这里分析的数据集中,可以观察到viSNE岛和FlowSOM元簇之间的良好相关性(图3B)。为了快速识别每个元簇的表型,可以通过聚类建立FlowSOM元簇的热图视图(图3C)。
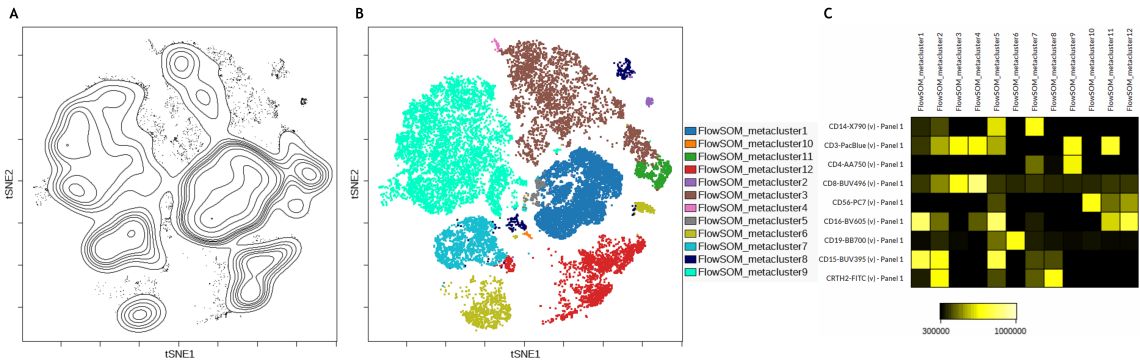 图3。使用viSNE、FlowSOM和热图显示进行子集识别。A) viSNE图的等高线图B)以FlowSOM元聚类为叠加图形尺寸的viSNE图C)FlowSOM元聚类标记表达的热图可视化。使用Kaluza分析软件对数据进行补偿和逻辑转换,并通过Kaluza Cytobank插件上传至Cytobank平台。viSNE在3个样本的11个群体定义标记上运行,共3000次迭代,30次困惑和0.5θ。FlowSOM设置为12个元集群和121个具有层次一致性集群的集群。绘图仅用于说明目的。
图3。使用viSNE、FlowSOM和热图显示进行子集识别。A) viSNE图的等高线图B)以FlowSOM元聚类为叠加图形尺寸的viSNE图C)FlowSOM元聚类标记表达的热图可视化。使用Kaluza分析软件对数据进行补偿和逻辑转换,并通过Kaluza Cytobank插件上传至Cytobank平台。viSNE在3个样本的11个群体定义标记上运行,共3000次迭代,30次困惑和0.5θ。FlowSOM设置为12个元集群和121个具有层次一致性集群的集群。绘图仅用于说明目的。
按通道着色的点图功能,根据数据集中通道上的强度为viSNE地图中的每个事件着色,可用于显示地图中的点彼此相邻的原因,或viSNE岛内事件之间的标记表达模式相似。图4显示了viSNE图上CD19、CD4和CD8的标记表达,并与FlowSOM元聚类进行了比较。

图4。viSNE图上亚群标记物的表达水平。A) CD19表达B)CD4表达C)CD8表达D)以FlowSOM元簇作为重叠图形维度的viSNE图。使用Kaluza分析软件对数据进行补偿和逻辑转换,并通过Kaluza Cytobank插件上传至Cytobank平台。viSNE在Cytobank插件上运行,对3个样本的11个群体定义标记进行3000次迭代、30次困惑和0.5θ。FlowSOM设置为12个元集群和121个具有层次一致性集群的集群。绘图仅用于说明目的。
图5。3个样本的比较。A) FlowSOM聚类覆盖在viSNE图上,箭头表示亚聚类1,星号表示亚聚类12b)CD16表达C)CD56表达。使用Kaluza分析软件对数据进行补偿和逻辑转换,并通过Kaluza Cytobank插件上传至Cytobank平台。viSNE在细胞库上对11个群体定义标记物进行了检测,viSNE对3个样本的所有CD45+白细胞进行了检测,共检测了3000次、30次、0.5θ。FlowSOM设置为12个元集群和121个具有层次一致性集群的集群。使用Kaluza分析软件对数据进行补偿和逻辑转换,并通过Kaluza Cytobank插件上传至Cytobank平台。在Cytobank平台上进行了进一步的数据分析。绘图仅用于说明目的。
结合viSNE和FlowSOM可以在样本之间进行定性比较,通过可视化viSNE图上特定标记物的表达可以增强定性比较(图5)。对比显示,被确定为亚群1(图5A,蓝色,箭头)的CD16+群体在样本G中显著,但在样本B和F中几乎不存在。它还显示样本F的亚群12中有大量CD56亮细胞(图5A,红色;星号)。
另一种可用于识别相似细胞组的无监督算法是SPADE。SPADE代表“密度标准化事件的生成树级数分析”4。SPADE将表型相似的细胞聚集成一个层次结构,允许对异质样本进行高通量、多维分析(图6)。可以添加气泡以将用户定义的总体阈值分配给SPADE发现的各种计算总体(集群)。

图6.2个样本的比较,CD16表达着色的黑桃树。使用Kaluza Analysis Software进行数据补偿和逻辑转换,通过Kaluza Cytobank Plugin上传到Cytobank平台。SPADE在11个群体上运行,向下取样至10%和50个节点。绘图仅供说明之用。
通过扩展手动门控策略,可以获得相应白细胞群的更深层次的免疫特征。同样,这通常基于表达模式的先验知识。图7提供了CD4+T细胞子集的示例。
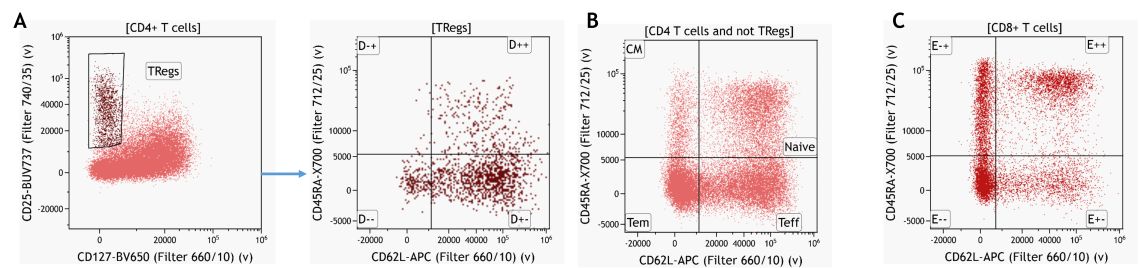
图7。知识驱动的调节性T细胞及其亚群鉴定。(A) CD4 T细胞记忆亚群门控于CD4+T细胞,不包括Treg(B)和CD8+T细胞记忆亚群。使用Kaluza分析软件分析数据。绘图仅用于说明目的。
样本之间的比较可以通过比较图或统计结果以及利用叠加功能或Kaluza比较图来完成(图8)。这种方法通常也以对可能的差异的假设为指导。
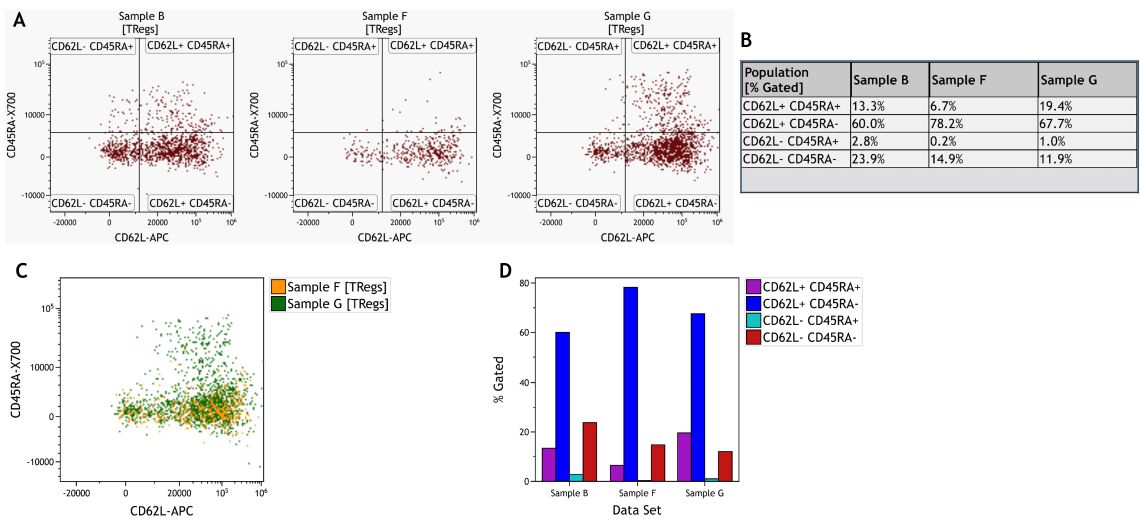
图8。样本间Treg亚群的比较。A) 每个样本的单个点图。B) 每个样本和子集的统计结果信息表。C) 两个样本的点图叠加。D) 比较图显示每个子集和样本的选通百分比。使用Kaluza分析软件对数据进行分析。绘图仅用于说明目的。
对于T细胞亚群的无监督鉴定,使用CD3+T细胞作为输入群体进行viSNE分析。图9a显示了viSNE图上CD4和CD8的表达。按照与之前相同的手动门控方法(见图7),使用pan T细胞上的象限门识别不同的CD45RA和CD62L表达模式,并在viSNE图上显示(图9b)。最后,使用FlowSOM进行分层一致性聚类,以识别10个元聚类(图9c)。人工选通和无监督聚类都可以识别相似的群体。
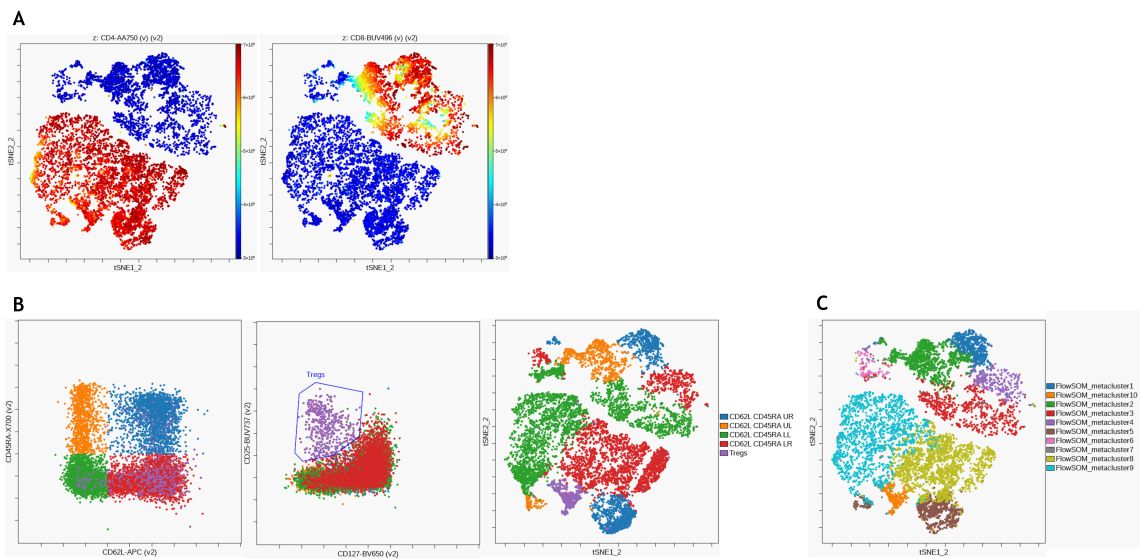
图9。T细胞亚群的无监督分析。(A) viSNE在Cytobank平台上进行了2000次迭代,结果viSNE图(B)显示了50和CD4(左)和CD8(右)的复杂表达。CD62L和CD45RA表达模式(左)以及调节性T细胞(中)通过手动选通和覆盖在viSNE图上的群体进行识别。(C) FlowSOM聚类使用标准化数据的分层聚类来检测100个聚类和10个元聚类。元聚类显示在viSNE地图上。使用Kaluza分析软件对数据进行补偿和逻辑转换,并通过Kaluza Cytobank插件上传至Cytobank平台。在Cytobank平台上进行了进一步的数据分析。绘图仅用于说明目的。
图10显示了使用手动选通和使用FlowSoM的无监督聚类识别CD8+内存子集的比较。
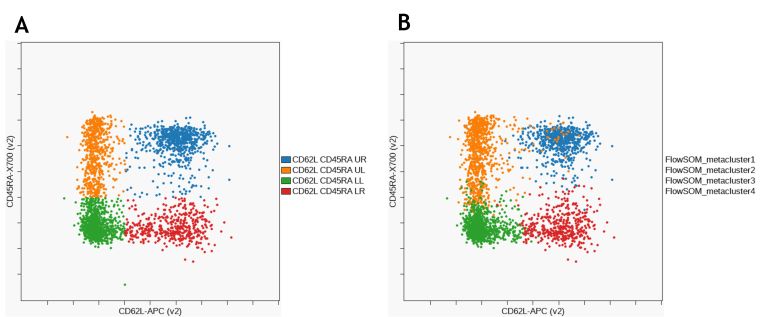
图10。人工门控法鉴定的CD8+ T细胞记忆亚群的比较(A)和FlowSOM (B)。使用Kaluza Analysis Software对数据进行补偿和逻辑转换,并通过Kaluza Cytobank Plugin上传到Cytobank平台。在Cytobank平台进行进一步的数据分析。绘图仅供说明之用。
总结
使用CytoFLEX LX上获得的20标记面板获得的3名供体数据用于显示一种手动门控策略,以识别白细胞亚群,并对T细胞亚群进行更深层次的分析。文中展示了利用viSNE在二维viSNE地图上可视化高维数据,并讨论了使用viSNE和SPADE来比较样品。最后,将FlowSOM自动聚类识别与人工浇注的结果进行了比较。viSNE、FlowSOM和SPADE等机器学习工具有助于可视化高参数数据和无偏性识别细胞子集。
成功的技巧
有关使用Kaluza分析软件的详细说明,请参阅Kaluza IFU C10986,有关使用Cytobank平台的详细说明,请访问support.Cytobank.org。本文件不代替使用说明。
关于此处执行的分析的更深入的讨论,请参见“使用机器学习算法探索高维流式细胞仪数据的潜力CytoFLEX LX上的20色面板示例“和”利用Kaluza和Cytobank平台的联合力量“技术笔记。
工具书类
- Amir ED,戴维斯吉隆坡,塔德莫医学博士,等. viSNE使高维单细胞数据可视化,并揭示白血病的表型异质性。自然生物技术。2013;31(6):545-552. 内政部:10.1038/nbt.2594。
- Van Gassen S、Callebaut B、Van Helden MJ、,等. FlowSOM:使用自组织图对细胞仪数据进行可视化和解释:FlowSOM。流式细胞术。2015;87(7):636-645. 内政部:10.1002/cyto.a.22625。
- Ortolani C.抗原。载:血液系统恶性肿瘤的流式细胞术。约翰·威利父子有限公司;2011:1-157。doi:10.1002/9781444398069.ch1。
- 邱鹏,西蒙兹EF, Bendall SC,等.使用SPADE从高维细胞仪数据中提取细胞层次结构。自然生物技术。2011;29(10):886-891。doi:10.1038/nbt.1991。
仅供研究使用,不用于诊断程序。
